搜索


由於抗生素的不合理使用,細菌耐藥性急速加劇。碳青黴烯類抗生素抗性在腸桿菌科細菌中的快速播散更是成為全球面臨的最嚴重公共衛生問題之一。耐藥基因常借助質粒、插入序列、轉座子👨🏼🏫、原噬菌體和基因組島等可移動遺傳元件在細菌中橫向轉移。這些元件序列組成了可移動基因組(mobile genome🧚🙍🏿♂️,mobilome),約占整個細菌基因組的20-30%。可移動基因組數據呈現出類別多🐔、結構復雜、語義標識多樣等異質性特點,如何解讀海量的測序數據是目前細菌基因組序列分析的主要瓶頸之一,急需一系列新工具和專業數據庫🏸。
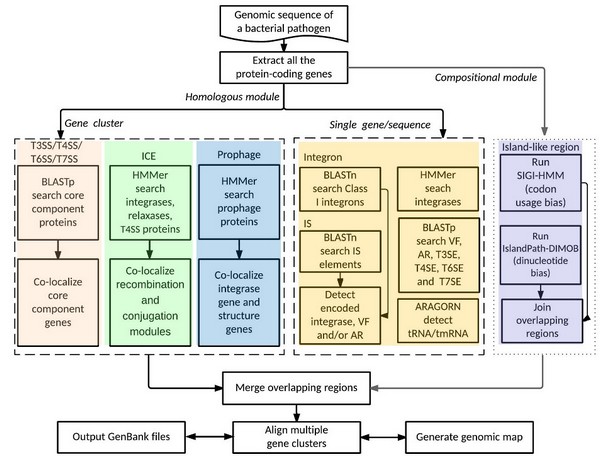
目前細菌基因組島的識別軟件主要采用了GC含量🎰、二核苷酸分布和密碼子使用等序列特征變量👢;VRprofile軟件則提出了“功能模塊為主⛪️,序列特征為輔”的新方法🦘,獲得了更高的預測精度。它在單基因和基因簇兩個水平上表征了基因組島、原噬菌體、跨膜分泌系統等長元件的核心組件💑,按不同種類元件設計了算法(圖1)。例如🦹🏽♀️,先用profile HMM查找特征蛋白同源基因🦿,再用基因簇共定位方法來識別島的整合和接合模塊。VRprofile在線工具的輸入界面支持全基因組序列或未註釋contigs;輸出界面可將識別結果和已知基因組島進行比對。此外,VRprofile服務器還提供了其他3個功能互補的在線軟件🧑🏿🦲:細菌基因組快速註釋工具CDSeasy,保守基因簇識別工具CGCfinder,COG基因集共定位工具COGviewer。
歐竑宇教授研究組長期從事微生物信息學研究🤽🏼♀️,系統性地開展了細菌可移動基因組數據庫構建🏚、軟件開發和實驗驗證工作。提出了用精細模塊化來剖析基因組島復雜數據的新方法,針對島的四模塊兩位點開發和維護了多個專業數據庫和軟件工具。目前🪆,該研究組正和浙江工業大學💐🫵🏽、美國密西根大學等合作夥伴一起➗,開發適用性更廣的新版本。VRprofile等新軟件的發展將為細菌耐藥性傳播機製的闡釋提供生物信息技術支撐🧑。該研究工作得到了國家自然科學基金和973項目的資助。


 首頁
首頁

 EON4概況
EON4概況

 師資隊伍
師資隊伍

 人才培養
人才培養

 招生就業
招生就業

 科學研究
科學研究

 平臺基地
平臺基地

 黨群工作
黨群工作

 校友之家
校友之家

 安全工作
安全工作

 網上辦事
網上辦事

 當前位置:
當前位置: